研 究 内 容
当研究室では、バイオインフォマティクス(生物情報科学)、計算科学、データサイエンスの手法を用いて、生体高分子の立体構造解析、疾患や薬理の分子構造論的探究、in silico 創薬に関する研究を進めています。
疾患に関連するタンパク質の立体構造解析
X線溶液散乱(SAXS)などの物理化学的手法と、束縛条件付きMD(分子動力学)法などの計算科学的手法を組合せて(→SAXS_MDによるデモ動画)、通常の方法(X線結晶解析、NMR、電子顕微鏡)では解析困難なタンパク質の立体構造を、迅速かつ簡便に解析する方法を開発し、実際に応用しています。
SAXSデータは、高エネルギー加速器研究機構(つくば)の放射光実験施設(Photon Factory)で測定します。また当研究室で独自に開発した解析・計算プログラムを、こちらで公開しています。

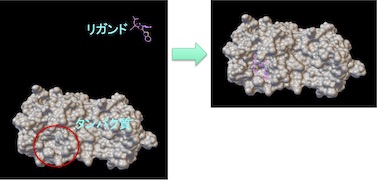
in silico 創薬
分子モデリング、分子動力学(MD)計算、定量的構造活性相関(QSAR)などの既存の確立された手法を、高速の汎用計算機(GPGPUワークステーション)で行うことにより、有用な医薬品候補分子をデザインします。さらに薬物有害反応(ADR)を検出するため、独自のInverse Virtual ScreeningシステムであるVOLTIS(Virtual Operation of Ligand and Target Interaction System)を構築しています。
またデータサイエンスと機械学習の手法を用いて、in silicoで薬物動態(ADME)や毒性を考慮した医薬品の設計を行います。特にウイルス感染症に対しては、結合ポケットの可変性を考慮したドッキングシミュレーションを行うことにより、変異ウイルスの標的とも結合できるリガンド分子の設計に取り組んでいます。
VOLTESプロジェクト
Protein Data Bankに登録されている立体構造に関するビッグデータを、位相幾何学、進化計算、AI(人工知能)の手法を併用してマイニングを行い、タンパク質の立体構造に関する新しい知見を得るとともに、分子の論理的構造設計などに応用するVOLTES(Virtual Optimization of Local TErtiary Structures)プロジェクトを進めています。現在VOLTES1、VOLTES-MCS、VOLTES2が完成し、タンパク質のトポロジー解析やリバースエンジニアリングに活用しています。またVOLTES-DB、VOLTES-AIを開発中です。

卒論生・大学院生に対する研究指導
当研究室では、全員がPythonの十分なプログラミングスキルを習得するとともに、研究テーマに応じて他のプログラミング言語の習得を勧めています。またプログラミング作法だけでなく、数値計算の基礎(桁落ちや丸め誤差)、手続きやデータの抽象化など、その背景になる計算機科学の原理や言語理論(構文解析や計算可能性)についても指導しています。
さらに大学院生は、研究室で独自に作成した教材を用いた研修プログラムを受講したうえで、グラフ畳み込みネットワーク(GCN)や相関ルールマイニングなどの手法を学習し、バイオに強いAI技術者としての素養を身に付けていきます。
最近のトピックから
当研究室は、愛知県医療療育総合センター発達障害研究所の河内全博士との共同研究により、先天性口唇口蓋裂の原因遺伝子PARG1(ArhGAP29)タンパク質の基質認識機構や変異の影響を構造計算科学の視点で明らかにするために、触媒活性を担う立体構造を推定し、触媒ドメインに存在する点突然変異が基質との結合に及ぼす影響を複合体の分子動力学解析によって明らかにしました(論文1)。
SAXS(X線溶液散乱)は、単分散データの解析を前提としますが、実際の測定試料では凝集体の形成が避けられない状況にしばしば遭遇します。当研究室客員研究員の松村博士は、SAXSの多分散データから抽出した単分散成分に対して、粗視化構造モデルを構築したところ、単分散データから構築したモデルとよく一致し、本研究の手法が、SAXSデータの補助的な解析手法として有効なことを示しました(論文2)。
最近の機械翻訳の精度向上は著しく、特に生成AIによる自動翻訳ではテキストのジャンルを的確に判断したうえで、原文の文法構造と単語の意味に関する限りほぼ間違いなく再現してくれるようになりました。ただし訳文は英文の構造に束縛されて、翻訳調のぎこちない日本語になっていることが多いため、当研究室では東京家政大学の甲斐基文教授との共同研究により、これをさらに、日本語のnative speakerにとって自然な表現、初めから英文なしで日本語で書いたときに現れるような文に変換するための方法論を開発し(論文3)、実際の書籍の翻訳に応用しました。
当研究室で開発したプログラムVOLTESは、タンパク質の立体構造とトポロジー情報を二面角の「木」で表し、入れ子のリストとして実装しています。VOLTESを用いたリバースエンジニアリングの手法により、1アミノ酸残基の置換が、分子構造上離れた部位に及ぼす影響について合理的に説明しました(論文5)。本件については、近畿大学・藤田医科大学・京都大学と共同プレスリリースを行いました。また当研究室の竹内は、VOLTESに特化したGCNを設計し、酵素分子の立体構造とトポロジー情報からEC番号の推定を機械学習で行うことにより、その有効性を実証しました。以上の成果は、情報処理学会第86回全国大会で発表しました。